
发布日期:2024-04-09浏览次数:669来源:蓝景科信
技术简介
Ribo-seq,又称为Ribosome Profiling或者翻译组测序,能够对与核糖体结合并正在被翻译的约30 nt的mRNA片段进行测序,详细检测体内的翻译状态,Ribo-seq是连接转录组学与蛋白质组学之间的桥梁。该技术可构建癌细胞全基因组水平上蛋白翻译图谱,量化蛋白质翻译效率,为基因表达转录后调控研究提供支持。
2016年发表在Cell上的“Modulated Expression of Specific tRNAs Drives Gene Expression and Cancer Progression”文章,发现了转移性乳腺癌细胞中表达上调的特异性tRNA,这些tRNA通过增强富含同源密码子的转录本的稳定性和翻译来驱动癌细胞转移。这篇文章主要通过三个关键证据支持研究者的观点:
l 特异性tRNA(tRNAGluUUC和tRNAArgCCG)在高转移性乳腺癌细胞中表达上调。
l tRNAs促进了富含其同源密码子的转录本的稳定性和翻译。
l tRNAGluUUC通过直接上调EXOSC2和增强GRIPAP1来驱动转移。
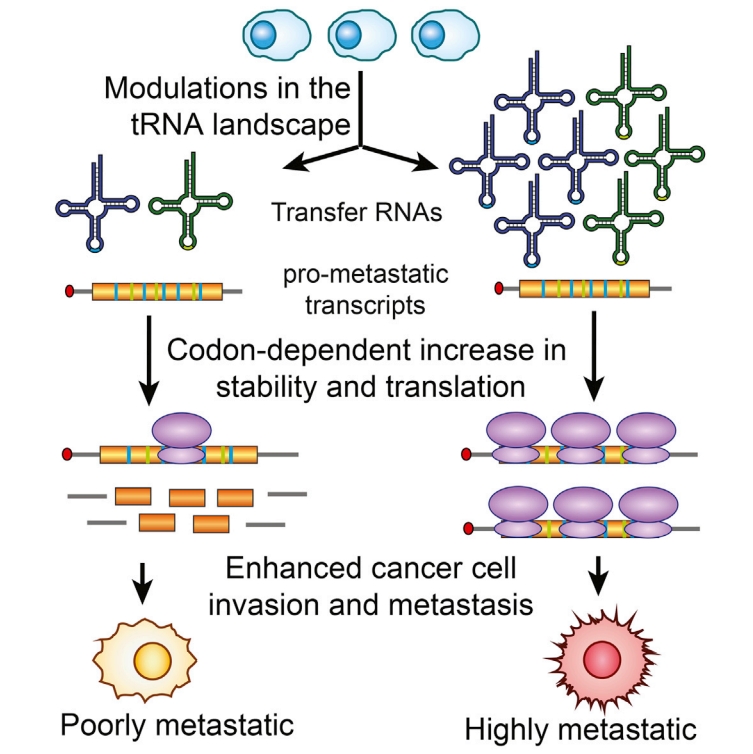
在之前的研究中,科学家发现编码序列中存在特异性密码子偏好,影响了蛋白质的表达水平。另一方面,在不同物种中,tRNA含量与蛋白质合成时密码子使用偏倚相关,并调节基因的表达。这些研究将细胞通路激活与tRNA含量联系起来。这篇文章的研究目的是通过对比非致瘤细胞系和乳腺癌细胞系中tRNA丰度,揭示tRNA的调节效应以及在肿瘤进程中发挥的作用。
研究者首先对不同细胞系(非致瘤上皮细胞系MCF10a,低转移乳腺癌细胞系MDA-231和CN34,以及相对应的高转移亚系MDA-LM2和CN-LM1a)的tRNA进行测序和定量,发现两组低转移乳腺癌细胞为获得更高的转移能力而选择相似的tRNA丰度调节模式,并证实了特异性tRNA(tRNAArgCCG和tRNAGluUUC)在高转移性乳腺癌细胞中表达上调,验证了tRNAs表达上调后癌细胞转移和侵袭能力增加(但与细胞增殖能力无关),进一步支持了tRNA水平调节可以特异性促进癌细胞转移的观点。
研究使用Ribo-seq技术分别对对照组(Control)、tRNAArgCCG过表达组(tRNAArg-OE)和tRNAGluUUC过表达组(tRNAGlu-OE)细胞系进行分析(Ingolia et al., 2014),检测活跃核糖体翻译组的调控结果,用于评估tRNA上调对转录后调控过程的影响。通过这些数据,科学家观察到了普遍存在的核糖体保护片段(RPFs,ribosome protected fragments)的周期性和长度(Figures 4A, 4B, S4B, and S4C)。在过表达细胞系中,一些基因的相对核糖体占有率较高,这些基因的编码序列中富含与特异性tRNA配对的同源密码子(Figures 4C and S4D)。根据上述结果,测量了约4000个蛋白质的表达水平直接评估tRNA调节对蛋白质表达的影响。在过表达细胞系中,与特异性tRNA同源的密码子含量较高的转录本普遍稳定。
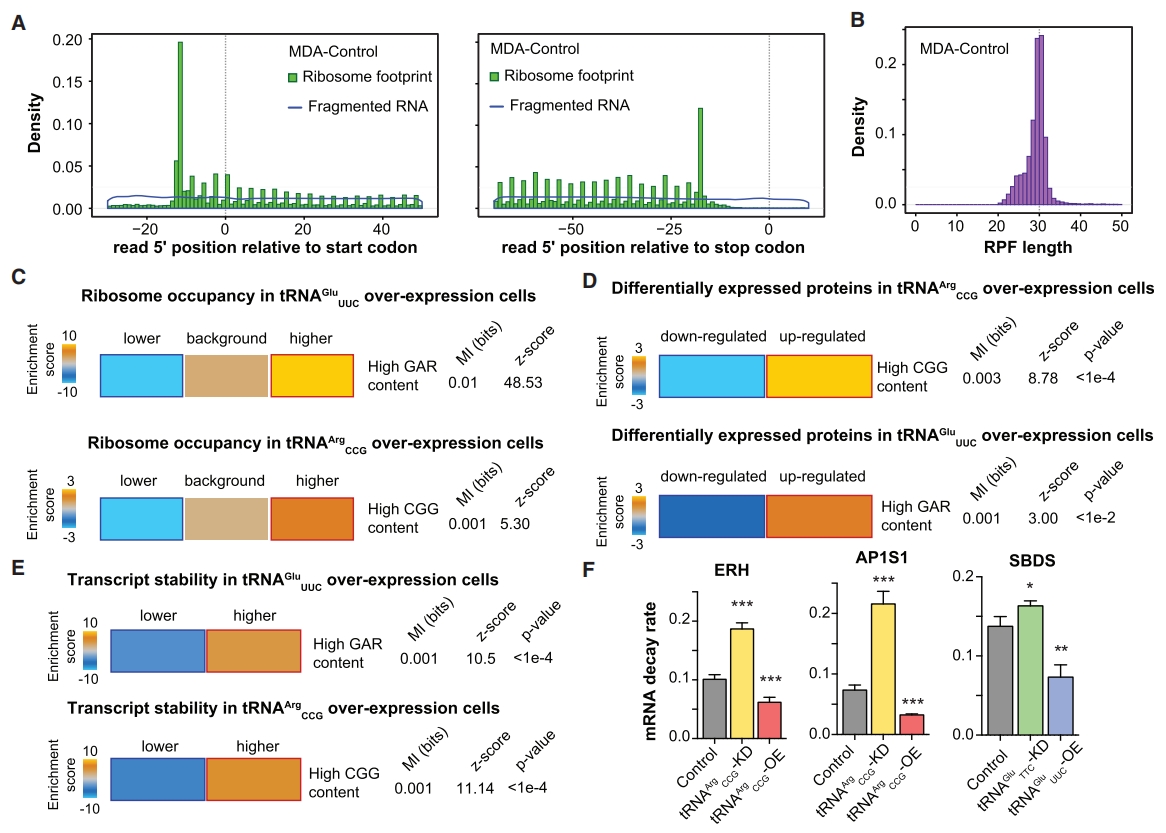
Figure 4. Post-Transcriptional Consequences of tRNAGluUUC and tRNAArgCCG Upregulation
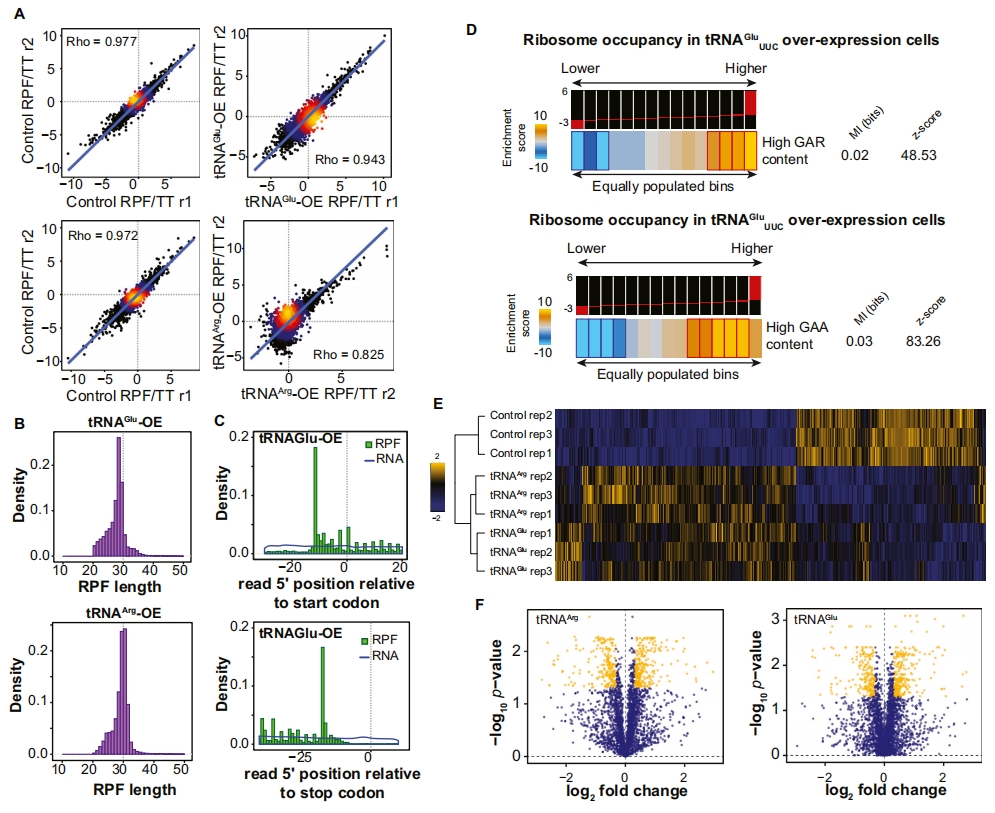
Figure S4. tRNAGluUUC and tRNAArgCCG Overexpression Alters Ribosomal Occupancy and Translational Landscapes, Related to Figure 4
该研究分析了全基因组数据并重点关注了tRNAGluUUC过表达细胞系的核糖体谱数据,确定了当tRNAGluUUC丰度更高时,EXOSC2和GRIPAP1是潜在靶点。这些基因在高转移细胞中高表达,但转录水平上无显著上调,表明这些基因上调主要是由转录后调节。
利用Ribo-seq技术分析核糖体保护片段(RPF),将tRNA偏好、核糖体占用和蛋白质表达之间相关联。为了量化tRNA含量的变化对活跃核糖体翻译组和蛋白质表达的影响,研究者定义了一个tRNA偏好评分参数,并对转移不良亲本细胞系(MDA-par和CN34-per)及其高转移衍生系(MDA-LM2和CN34-LM1a)进行了分析。核糖体保护片段(RPF)被归一化为每个细胞系的总RNA(TT),用于解释基因表达的差异。亲本与衍生系之间的RPF/TT比率在生物学重复中保持一致,显著正相关(Figures 7A and S7A)。将每个基因的tRNA偏好评分与其各自矫正的核糖体足迹重叠后,发现具有较高tRNA偏好评分的转录本在那些受核糖体结合较多的转录本中强烈富集(Figure 7B)。研究者随后将每个基因转录本丰度进行归一化处理,证实了高转移亚系中,tRNA偏好得分高的基因显著富集(Figure 7C)。以上结果表明,差异tRNA表达为蛋白质翻译图谱的改变提供重要信息。

Figure 7. tRNA Preference Scores Were Informative of Differential Ribosome Occupancy and Protein Expression
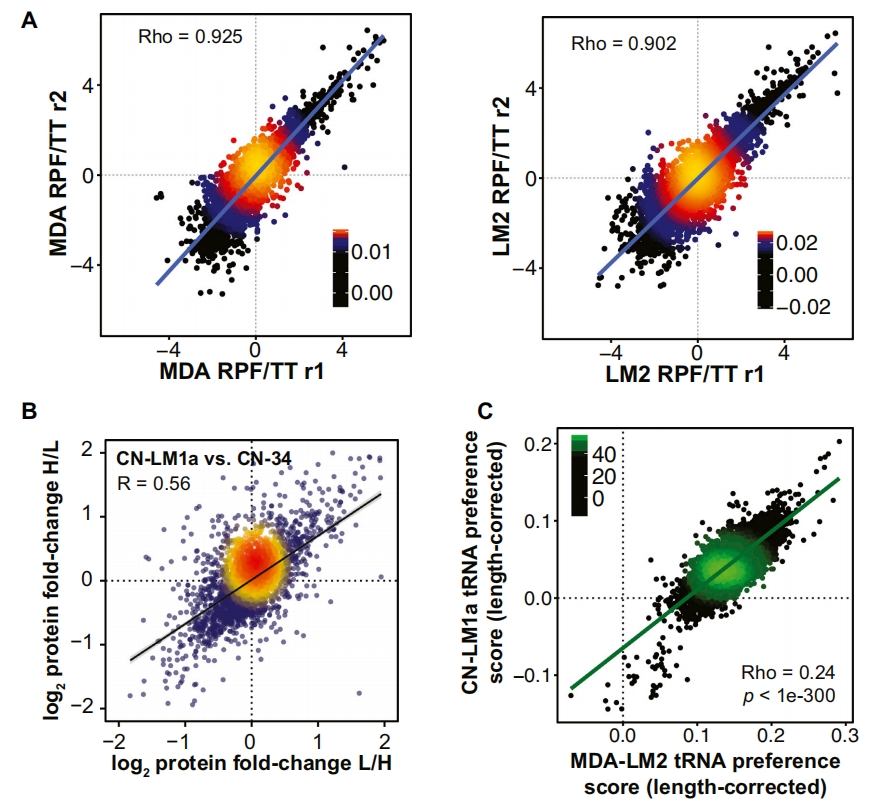
Figure S7. Differential Protein Expression and tRNA Preference Scores, Related to Figure 7
这项研究证实了tRNA丰度的改变可以调节细胞中蛋白的表达,并且癌细胞可以通过调节tRNA水平来调整癌症进程中的多数启动子的表达。虽然文章中这种tRNA分析方法是基于乳腺癌细胞系研究,但研究者认为在其他疾病、模型和物种中还有更广阔的应用前景。
小结
Ribo-seq技术检测到核糖体结合的转录本与tRNA表达显著相关,将基因表达调控中的转录过程与翻译过程关联起来,可以通过检测mRNA核糖体保护片段(RPF),识别翻译过程中的tRNA调控事件。Ribo-seq技术可以与DAP-seq/ChIP-seq、RNA-seq、tRNA-seq和质谱等技术一起,将基因表达的调控过程完整呈现出来。
引用文献
Goodarzi H, Nguyen HCB, Zhang S, Dill BD, Molina H, Tavazoie SF. Modulated Expression of Specific tRNAs Drives Gene Expression and Cancer Progression. Cell. 2016. 165(6):1416-1427.