
发布日期:2025-07-11浏览次数:517来源:蓝景科信
Ribo-seq简介
Ribo-seq(Ribosome Profiling,核糖体印迹测序,翻译组测序),通过识别与核糖体结合的mRNA,对正在翻译的RNA片段(即核糖体保护片段,Ribosome Protect Fragments, RPFs)进行捕获和测序分析。其核心原理是利用低浓度RNase处理核糖体-新生肽链复合物时,未被核糖体覆盖的mRNA会被降解,而被核糖体保护的约28~30bp的RNA小片段,即核糖体足迹(ribosome footprints,RFP)得以保留。
Ribo-seq技术的革命性突破在于它能够通过高通量测序解析RPFs的位置和序列,可精确定位活细胞中正在翻译的核糖体在RNA上的具体位置,并可帮助研究人员推断起始密码子、识别上游开放阅读框(uORFs)、解析密码子翻译动态等信息。
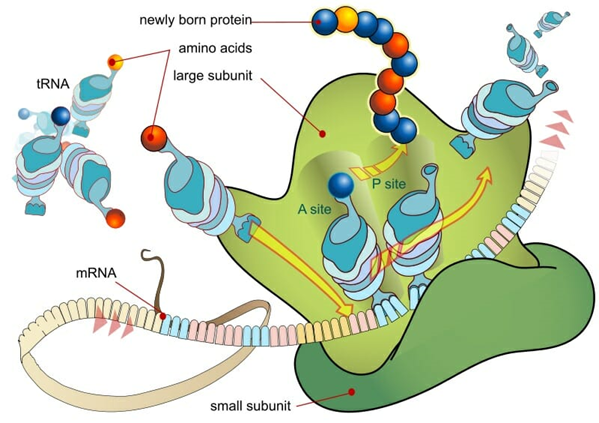
在神经科学领域,基因表达调控研究长期聚焦于转录水平,然而越来越多的证据表明,翻译调控在突触可塑性、神经元分化及认知记忆形成中扮演关键角色(例如,神经元受刺激后突触局部的mRNA翻译动态直接影响突触强度变化)。作为连接转录组与蛋白质组的桥梁,Ribo-seq精准揭示了神经系统中蛋白质合成的调控机制。
Ribo-seq研究思路
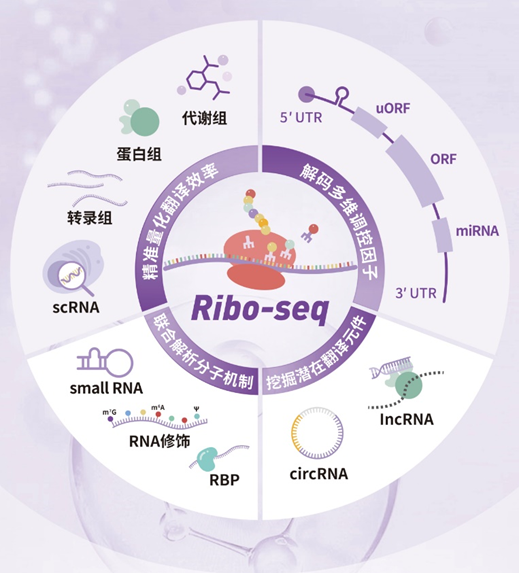
通过系统梳理Ribo-seq相关研究文献,我们将其核心研究思路归纳为四大方向:精准量化翻译效率,解码多维调控因子,挖掘潜在翻译元件,联合解析分子机制(如下图所示),本期推文我们将分别介绍这四种研究思路及其在神经科学方向的应用。
一:精准量化翻译效率:破解“转录-翻译”脱钩之谜
Ribo-seq通过捕获核糖体保护的mRNA片段(RPFs),与RNA-seq数据联合分析可计算基因的翻译效率(Translational Efficiency, TE),计算公式为:TE=Ribo-seq丰度/RNA-seq丰度。这一指标直接反映了单位mRNA的翻译活性,是基因在翻译水平调控强度的直观体现。
TE的独特价值在于它能揭示不依赖于转录水平的翻译调控——即使mRNA总量不变,通过调控核糖体结合效率(如eIF2α磷酸化抑制翻译起始),TE也可显著改变蛋白质合成量。这种“翻译效率调控”正是解答“基因如何在翻译水平实现精准表达调控”的核心钥匙,例如在神经元受刺激后,突触局部mRNA的TE可在分钟内激增,快速响应神经信号而不依赖新基因的转录。
[文献案例] 通过Ribo-seq精准量化翻译效率,聚焦亨廷顿病(HD)中线粒体翻译缺陷的机制解析

文章标题:Ribosome Profiling and Mass Spectrometry Reveal Widespread Mitochondrial Translation Defects in a Striatal Cell Model of Huntington Disease
研究背景:亨廷顿病 (Huntington's disease, HD) 是一种由亨廷顿基因 (huntingtin, HTT) 中的多谷氨酰胺重复序列扩展引起的神经退行性疾病。该病以纹状体显著萎缩、认知和精神障碍以及舞蹈样运动为特征。线粒体是一个拥有半自主系统的细胞器,有自己独特的基因表达和mRNA翻译机制,其功能依赖于核基因组与线粒体基因组(mtDNA)的协同调控。人类线粒体基因组 (mtDNA) 包含37个蛋白编码基因,包括2个rRNA基因和22个tRNA基因,以及13个氧化磷酸化 (OXPHOS) 反应相关复合物 (I、III和IV) 的编码基因。而mtDNA维护、复制、转录、翻译、翻译后修饰、运输、组装和OXPHOS复合物(II和V)表达所需的基因均由细胞核编码。因此,这种双基因组调控模式使得线粒体功能的研究颇具挑战,特别是OXPHOS复合物的翻译和组装,因为这些复合物是由不同的基因组编码的。已有研究表明HD与纹状体线粒体功能障碍相关,但mtDNA编码mRNA的翻译效率在HD中的变化规律及调控机制尚不清楚。
该研究使用小鼠纹状体细胞模型,包括野生型HTT(Control)、杂合突变型HTT(HD-het)和纯合突变HTT(HD-homo)的细胞系。通过Ribo-Seq分析线粒体编码的mRNA转录本上的核糖体占用情况。结合TMT-MS技术对成熟线粒体蛋白组进行定量分析,旨在揭示HD中mtDNA翻译调控与线粒体功能障碍的因果关系。
研究发现:
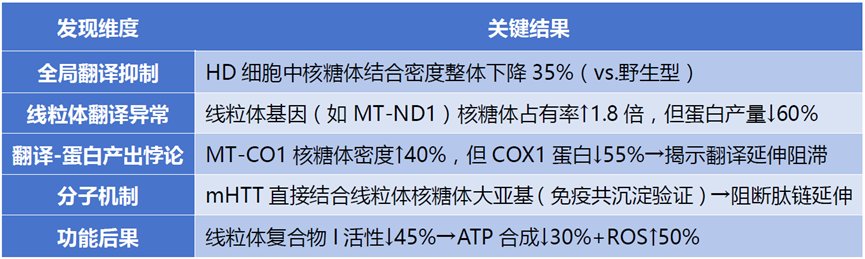
研究结论:该研究通过Ribo-seq/质谱双模态量化,首次揭示亨廷顿病中“核糖体滞留却蛋白荒废”的线粒体翻译悖论,提供了HD中线粒体翻译缺陷的证据,揭示了线粒体翻译调控的复杂性。发现的翻译失调可能对HD的发病机制有重要影响,为未来的治疗干预提供了新的视角。
二:解码多维调控机制:捕捉翻译“调速器”
通过TE(翻译效率)值,我们获知了基因的翻译调控变化。那是什么导致了基因的翻译调控发生显著变化?Ribo-seq可解析非经典翻译调控元件(如uORF、IRES、核糖体滞留位点)对翻译速率的影响,揭示RNA结构、RNA结合蛋白(RBPs)或修饰如何动态调控神经基因的翻译起始/延伸效率。
[文献案例] 精神分裂症的翻译刹车失灵:miR-1271-5p缺失引发突触蛋白失控合成

文章标题:Characterising the Transcriptional and Translational lmpact of the Schizophrenia-Associated miR-1271-5p in Neuronal cells
研究背景:作者在之前的miRNA-seq分析结果中发现,miR-1271-5p是一种与精神分裂症有关,且在神经元中表达的miRNA,但目前miR-1271-5p这一miRNA的作用机制并不明晰。
研究目的:利用RNA-seq、Ribo-seq研究miR-1271-5p在神经元中的作用机制和功能。
研究发现:

研究结论:miR-1271-5p作为翻译“刹车器”,通过结合靶基因3'UTR直接抑制核糖体起始(非降解mRNA),其失灵导致突触蛋白失控合成。该研究不仅揭示精神分裂症的翻译调控新机制,更提供了靶向RNA的精准干预思路。
三:挖掘潜在翻译元件:扫描基因组的“暗物质”
传统基因注释体系往往忽略非典型翻译事件,而Ribo-seq凭借其高分辨率的核糖体足迹分析,可在全基因组范围内揭示新型翻译元件:
sORF(小开放阅读框):多位于mRNA非编码区(如5’ UTR)或长链非编码RNA中,编码微蛋白(<100个氨基酸)。
circRNA(环状RNA):部分含内部核糖体进入位点(IRES)的circRNA可启动翻译,具有翻译潜力。
lncRNA(长链非编码RNA):传统定义为非编码RNA,但Ribo-seq证实部分lncRNA含隐蔽sORF。
这些曾被视为“基因组暗物质”的翻译元件,在神经系统中可能具有特殊功能,参与神经发育、突触可塑性和神经退行性过程。
[文献案例] 人脑翻译组图谱破译:38,000个隐藏微蛋白揭示神经发育新维度

文章标题:Developmental Dynamics of RNA Translation in the Human Brain
研究背景:基因表达调控对神经发育、可塑性及认知功能至关重要,虽已有研究剖析发育中人脑的转录情况,但对伴随的翻译调控认知存在缺口。核糖体分析显示,酵母、心脏及肿瘤组织中先前未知的小开放阅读框(sORFs)可编码微蛋白并发挥重要调控作用,然而类似微蛋白在发育中人脑的性质和作用几乎未被表征。
研究材料:30个产前大脑皮质样本和43个成人大脑样本;人胚胎干细胞(hESC)衍生的神经元细胞
研究发现:

研究结论:该研究深入探讨了人类大脑发育过程中RNA翻译的动态变化。联合使用核糖体印迹测序(Ribo-seq)和RNA测序(RNA-seq)技术绘制了人类大脑的翻译图谱,揭示了基因表达调控的关键节点,并识别了数千个之前未知的翻译事件,包括产生人类大脑特异性微蛋白(microproteins)的小开放阅读框(sORFs)。
四:联合解析分子机制:整合多组学破解翻译调控的因果链条
Ribo-seq的强大功能在与其他组学技术整合时更为显著:
翻译组+表观转录组(如 m6A-seq):揭示RNA修饰对翻译的动态调控
翻译组+蛋白质组(TMT-MS/Label-free):不仅能验证翻译效率(TE)与蛋白质丰度的一致性,更能发现翻译后调控的 “盲区”
翻译组+代谢组(LC-MS 代谢物检测):探索翻译重编程与代谢适应
这种多维整合策略为解析神经疾病的复杂机制提供了系统视角。
[文献案例] 破解脆性X综合征蛋白的翻译调控密码,揭示YTHDF1调控mRNA翻译过程的“开关”—FMRP磷酸化

文章标题:FMRP phosphorylation modulates neuronal translation through YTHDF1
研究背景:脆性X染色体综合症(FragileXsyndrome,FXS)是常见的遗传性智力障碍疾病。FXS的病因主要是编码脆性X染色体智力低下蛋白(FMRP)的FMR1基因5’端非翻译区CGG重复片段的增多,导致FMR1基因沉默。FXS的重要特征之一是大脑中非正常活跃的mRNA翻译。FMRP通常被认为是mRNA翻译的抑制因子。
该研究阐明了一条在神经元细胞内YTHDF1翻译功能受FMRP磷酸化调控的路径,发现了YTHDF1可作为治疗FXS的潜在药物靶标,并揭示了中药丹参重要成分丹酚酸C(SalvianolicacidC,SAC)可通过抑制YTHDF1,进而缓解FXS疾病症状的新策略。
研究发现:

研究结论: FMRP缺失→YTHDF1持续激活→阻断YTHDF1-mRNA互作(m6A⁺基因TE↑)→翻译抑制→突触可塑性失衡→脆性X综合征表型。
该研究首次揭示了FMRP磷酸化作为“翻译开关”通过调控m6A“阅读子”YTHDF1功能影响突触可塑性的机制,同时表征了YTHDF1相分离相关的相互作用蛋白,提出YTHDF1与核糖体相互作用并且形成翻译活性高的凝聚体。通过发现的这一调控路径,他们指出YTHDF1是FXS里的一个新的药物靶点,并且表征了一个YTHDF1特异性的小分子抑制剂。这为进一步理解YTHDF1在不同生物学系统中的功能调控以及FXS的药物研发提供了新的方向和分子基础。
结语
以上就是Ribo-seq在神经科学方向的应用介绍。通过梳理这些研究案例,我们不难发现Ribo-seq在神经科学中已形成成熟研究范式,从翻译效率动态量化到跨组学机制整合。熟悉Ribo-seq技术的伙伴们,可以充分借鉴这些成熟的研究范式和分析策略,应用于自己的研究项目中。
参考文献:
1. Dagar S, Sharma M, Tsaprailis G, Tapia CS, Crynen G, Joshi PS, Shahani N, Subramaniam S. Ribosome Profiling and Mass Spectrometry Reveal Widespread Mitochondrial Translation Defects in a Striatal Cell Model of Huntington Disease. Mol Cell Proteomics. 2024 Apr;23(4):100746.
2. Kiltschewskij DJ, Geaghan MP, Cairns MJ. Characterising the Transcriptional and Translational Impact of the Schizophrenia-Associated miR-1271-5p in Neuronal Cells. Cells. 2020 Apr 18;9(4):1014.
3. Duffy EE, Finander B, Choi G, Carter AC, Pritisanac I, Alam A, Luria V, Karger A, Phu W, Sherman MA, Assad EG, Pajarillo N, Khitun A, Crouch EE, Ganesh S, Chen J, Berger B, Sestan N, O'Donnell-Luria A, Huang EJ, Griffith EC, Forman-Kay JD, Moses AM, Kalish BT, Greenberg ME. Developmental dynamics of RNA translation in the human brain. Nat Neurosci. 2022 Oct;25(10):1353-1365.
4. Zou Z, Wei J, Chen Y, Kang Y, Shi H, Yang F, Shi Z, Chen S, Zhou Y, Sepich-Poore C, Zhuang X, Zhou X, Jiang H, Wen Z, Jin P, Luo C, He C. FMRP phosphorylation modulates neuronal translation through YTHDF1. Mol Cell. 2023 Dec 7;83(23):4304-4317.e8.