- 技术简介
- 技术流程
- CUT&Tag数据
- 技术对比
CUT&Tag(Cleavage Under Targets and Tagmentation)可以在全基因组范围内定位转录因子的结合位点或者组蛋白修饰发生的位置。与ChIP-Seq相比,CUT&Tag不需要甲醛交联和免疫共沉淀的过程,而是通过靶蛋白的抗体和Protein A的介导,使与Protein A融合的Tn5转座酶靶向并切割DNA,同时在序列两端加上测序接头,经PCR扩增后形成高通量测序文库。这项技术的数据信噪比高,重复性好。
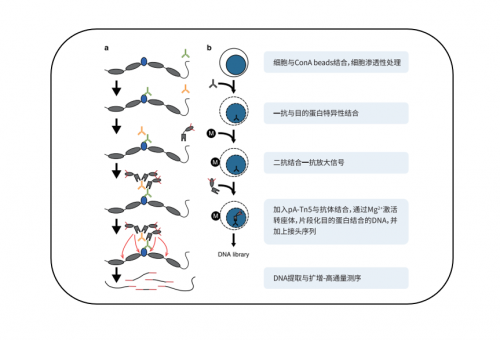
实验流程图

细胞与ConA beads结合——细胞渗透性处理——一抗与目的蛋白特异性结合——二抗结合一抗放大信号——加入pA-Tn5与抗体结合——通过Mg2+激活转座体,片段化目的蛋白结合的DNA,并加上接头序列——DNA提取与扩增-高通量测序
| 客户提供材料 | 交付结果 |
冻存细胞:细胞数>10万 冻存组织:组织量>40 mg 一抗(请使用ChIP级别的一抗,提供未稀释的抗体原液, 送样前请将抗体的说明书发给我们,以便安排后续实验) | 实验过程图 高通量测序原始数据 生物信息学分析结果 |


使用CUT&Tag,鉴定H3K27me3在染色质水平上的peaks。与ChIP-seq相比,在相同Reads条件下,两者的Peaks一致,并且CUT&Tag的信噪比更高、背景更低。
参考文献:
Kaya-Okur HS, Wu SJ, Codomo CA, Pledger ES, Bryson TD, Henikoff JG, Ahmad K, Henikoff S. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun. 2019 Apr 29;10(1):1930. doi: 10.1038/s41467-019-09982-5.
| CUT&Tag,DAP-seq,ChIP-seq技术对比 | |||
| 技术名称 | CUT&Tag | DAP-seq | ChIP-seq |
| 实验模式 | 体内 | 体外 | 体内 |
| 是否需要特异性抗体 | 是 | 否 | 是 |
| 样本适用性 | 对细胞活性要求高,不兼容冷冻样本,能够对极少量样品进行分析 | 各种组织器官,新鲜组织、冻存组织皆可 | 各种组织器官,新鲜组织、冻存组织皆可 |
| 信噪比 | 背景噪音低,所需测序深度少 | 噪音高,所需测序量相对较多 | 噪音高,所需测序量相对较多 |







